Repräsentierung und Visualisierung von Daten¶
Einleitung¶
Beim maschinellen Lernen geht es darum, Modelle an Daten anzupassen. Aus diesem Grund beginnen wir damit zu zeigen wie Daten dargestellt werden können, um vom Computer verstanden zu werden.
Zu Beginn dieses Kapitels haben wir Tom Mitchells Definition des maschinellen Lernens zitiert: Ein wohlgestelltes Lernproblem: Ein Computerprogramm soll aus der Erfahrung E in Bezug auf eine Aufgabe T und ein Leistungsmaß P lernen, wenn sich seine Leistung auf T, gemessen durch P, mit Erfahrung E verbessert. Daten sind der "Rohstoff" für maschinelles Lernen. ML lernt aus Daten. In Mitchells Definition sind "Daten" hinter den Begriffen "Erfahrung E" und "Leistungsmaß P" verborgen. Wie bereits erwähnt, benötigen wir gelabelte Daten, um unseren Algorithmus zu trainieren und zu testen.
Es empfiehlt sich jedoch, dass man sich mit seinen Daten vertraut macht, bevor man mit dem Training des Klassifikators beginnt.
Numpy bietet ideale Datenstrukturen zur Darstellung der Daten und Matplotlib bietet hervorragende Möglichkeiten zur Visualisierung der Daten.
Wie man hierbei vorgeht, wollen wir im Folgenden anhand der Daten zeigen, die sich im Modul sklearn befinden.
Ein einfaches Beispiel: das Iris-Datenset¶
Was war das erste Programm, das Sie gesehen haben? Ich wette, es könnte ein Programm gewesen sein, das "Hello World" in einer Programmiersprache ausgegeben hat. Höchstwahrscheinlich habe ich recht. Fast jedes Einführungsbuch oder Tutorial zur Programmierung beginnt mit einem solchen Programm. Es ist eine Tradition, die auf das Buch "The C Programming Language" von 1968 von Brian Kernighan und Dennis Ritchie zurückgeht!
Die Wahrscheinlichkeit, dass der erste Datensatz, den Sie in einem Einführungstutorial zum maschinellen Lernen sehen, der "Iris-Datensatz" ist, ist ähnlich hoch. Der Iris-Datensatz enthält die Messungen von 150 Blüten von Schwertlilien (englisch "iris") aus drei verschiedenen Arten: Als Beispiel für einen einfachen Datensatz sehen wir uns die von scikit-learn gespeicherten Irisdaten an. Die Daten bestehen aus Messungen von drei verschiedenen Irisblütenarten. Es gibt drei verschiedene Arten von Schwertlilien (Iris) in diesem speziellen Datensatz wie unten dargestellt:
Iris Setosa (Borsten-Schwertlilie)

Iris Versicolor (Verschiedenfarbige-Schwertlilie)

Iris Virginica

Der Iris-Datensatz wird häufig wegen seiner Einfachheit verwendet. Dieser Datensatz ist in scikit-learn enthalten.
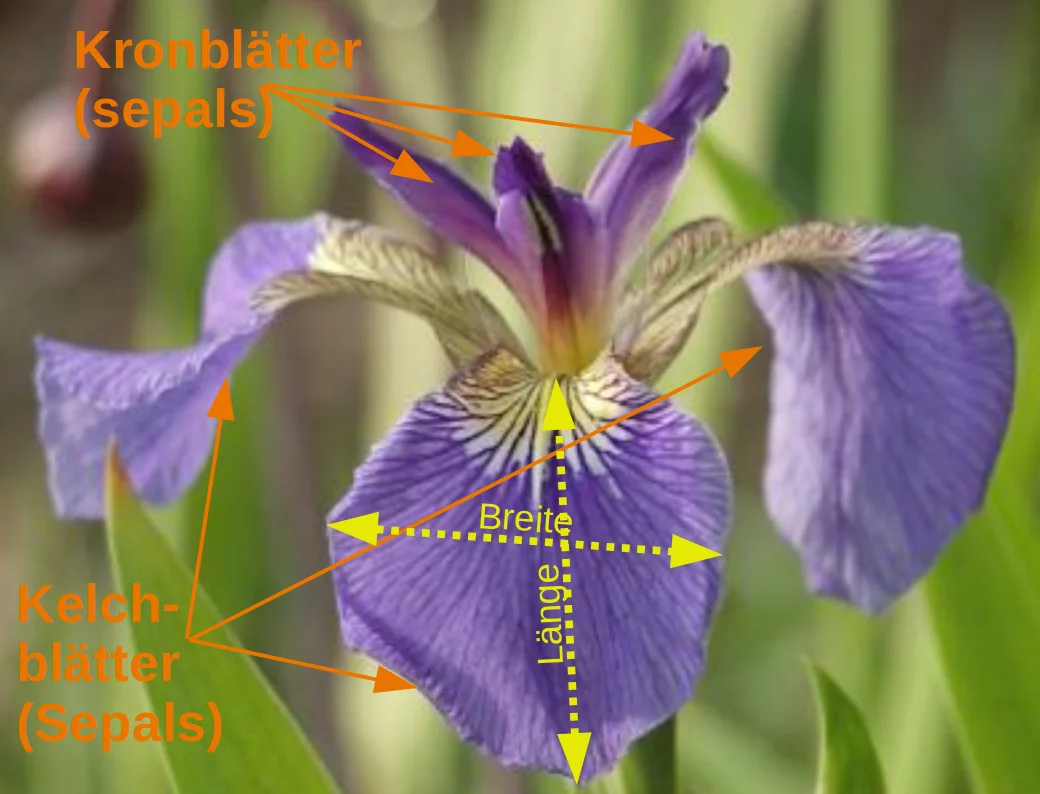
Die Daten bestehen aus den ermittelten Abmessungen der Kronblätter (Fachbegriff: Petalum oder engl. petal) und der Kelchblätter (Fachbegriff: Sepalum) von drei verschiedenen Lilienarten:
Merkmale im Iris-Datensatz:
- Kelchlänge (sepal length) in cm
- Kelchbreite (sepal width) in cm
- Kronblattlänge (petal length) in cm
- Kronblattbreite (petal width) in cm
Zu prognostizierende Zielklassen:
- Iris Setosa
- Iris Versicolour
- Iris Virginica
scikit-learn beinhaltet eine Kopie der Iris-CSV-Datei zusammen mit einer Hilfsfunktion, um diese Daten in numpy-Arrays zu laden:
from sklearn.datasets import load_iris
iris = load_iris()
Der resultierende Datensatz ist ein "Bunch"-Objekt:
type(iris)
The resulting dataset is a Bunch object: you can see what's available using
the method keys():
iris.keys()
Ein Bunch-Objekt ähnelt einem Dictionary, ermöglicht jedoch zusätzlich den Zugriff auf die Schlüssel im Attributstil:
print(iris["target_names"])
print(iris.target_names)
iris.data.shape enthält die Anzahl der Merkmale und Samples (Stichproben):
n_samples, n_features = iris.data.shape
print('Anzahl der Sampels:', n_samples)
print('Anzahl der Merkmale:', n_features)
# the sepal length, sepal width, petal length and petal width of the first sample (first flower)
print(iris.data[0])
Die Merkmale jeder Blume werden im Attribut data des Datensatzes gespeichert. Werfen wir einen Blick auf einige der Sampels:
# Blumen mit den Indizes 12, 26, 89 und 114
iris.data[[12, 26, 89, 114]]
Die Informationen über die Klasse jeder Probe, d.h. die Labels, werden im Attribut "target" des Datensatzes gespeichert:
print(iris.data.shape)
print(iris.target.shape)
print(iris.target)
import numpy as np
np.bincount(iris.target)
Mit der Bincount-Funktion von NumPy (oben) können wir sehen, dass die Klassen in diesem Datensatz gleichmäßig verteilt sind - es gibt 50 Blumen von jeder Art, wobei
- class 0: Iris-Setosa
- class 1: Iris-Versicolor
- class 2: Iris-Virginica
Diese Klassennamen werden im letzten Attribut gespeichert, nämlich target_names:
print(iris.target_names)
Daten in sklearn (scikit-learn)¶
Bei den Daten in scikit-learn kann man mit wenigen Ausnahmen davon ausgehen, dass sie als zwei-dimensionale Arrays abgespeichert sind. Ihre Gestalt (shape) ist von der Form "(n_samples, n_features)".
- n_samples: Die Anzahl der Samples: Jedes Sample ist ein zu verarbeitendes Objekt. Eine Stichprobe kann ein Dokument, ein Bild, ein Ton, ein Video oder ein astronomisches Objekt sein, Eine Stichprobe entspricht einer Zeile in einer Datenbank oder CSV-Datei, oder was auch immer man mit einem festen Satz von quantitativen Merkmalen beschreiben können.
- n_features: Die Anzahl der Merkmale oder besonderen Merkmale, die zur Beschreibung der einzelnen Merkmale verwendet werden können Artikel in quantitativer Weise. Features sind im Allgemeinen reelle Werte, können aber auch boolesche Werte oder Werte sein in einigen Fällen diskret bewertet.
Die Anzahl der Features muss im Voraus festgelegt werden. Es kann jedoch sehr hoch dimensioniert sein
(z.B. Millionen von Merkmalen), wobei die meisten von ihnen "Null" für eine gegebene Stichprobe sein können. Das ist ein Fall
wo scipy.sparse Matrizen nützlich sein können, sind sie
viel speichereffizienter als NumPy-Arrays.
Wie wir uns aus dem vorherigen Abschnitt (oder dem Jupyter-Notizbuch) erinnern, stellen wir Beispiele (Datenpunkte oder Instanzen) als Zeilen im Datenarray dar und speichern die entsprechenden Features, die "Dimensionen", als Spalten.
Die Daten für eine Blumen bestehen aus einer Zeile im Datenarray, und die Spalten (Merkmale) geben die Blumenmaße in Zentimetern an. Zum Beispiel können wir diesen Iris-Datensatz, bestehend aus 150 Beispielen und 4 Merkmalen, einem zweidimensionalen Array oder einer Matrix $\mathbb{R}^{150 \times 4}$ , im folgenden Format darstellen:
$$\mathbf{X} = \begin{bmatrix} x_{1}^{(1)} & x_{2}^{(1)} & x_{3}^{(1)} & x_{4}^{(1)} \\ x_{1}^{(2)} & x_{2}^{(2)} & x_{3}^{(2)} & x_{4}^{(2)} \\ \vdots & \vdots & \vdots & \vdots \\ x_{1}^{(150)} & x_{2}^{(150)} & x_{3}^{(150)} & x_{4}^{(150)} \end{bmatrix}. $$Der hochgestellte Index bezeichnet die i-te Zeile, und der tiefgestellte Index bezeichnet das j-te Merkmal.
Im Allgemeinen haben wir $n$ Zeilen und $k$ Spalten:
$$\mathbf{X} = \begin{bmatrix} x_{1}^{(1)} & x_{2}^{(1)} & x_{3}^{(1)} & \dots & x_{k}^{(1)} \\ x_{1}^{(2)} & x_{2}^{(2)} & x_{3}^{(2)} & \dots & x_{k}^{(2)} \\ \vdots & \vdots & \vdots & \vdots & \vdots \\ x_{1}^{(n)} & x_{2}^{(n)} & x_{3}^{(n)} & \dots & x_{k}^{(n)} \end{bmatrix}. $$print(iris.data.shape)
print(iris.target.shape)
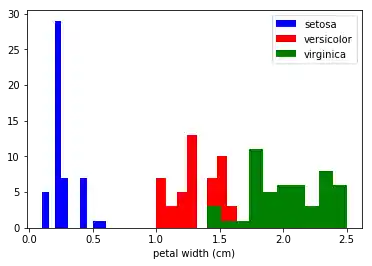
Visualisierung der Merkmale des Iris-Datensatzes¶
Diese Daten sind vierdimensional, aber wir können eine oder zwei der Dimensionen visualisieren zu einem Zeitpunkt mit einem einfachen Histogramm oder Streudiagramm.
Histograms of the features¶
import matplotlib.pyplot as plt
fig, ax = plt.subplots()
x_index = 3
colors = ['blue', 'red', 'green']
for label, color in zip(range(len(iris.target_names)), colors):
ax.hist(iris.data[iris.target==label, x_index],
label=iris.target_names[label],
color=color)
ax.set_xlabel(iris.feature_names[x_index])
ax.legend(loc='upper right')
fig.show()
Übung¶
Im obigen Code haben wir x_index auf 3 gesetzt, das bedeutet, dass wir uns das Histogramm der Breite der Kronblätter (petals) erzeugt haben. Schauen Sie sich entsprechend die Histogramme der anderen Größen von den Kelch- (sepals) und Kronblättern an.
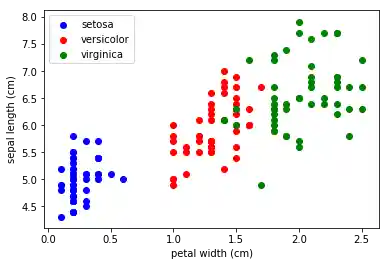
Streudiagramm mit zwei Merkmalen¶
Das nächste Diagramm zeigt zwei Merkmale in einem Diagramm:
import matplotlib.pyplot as plt
fig, ax = plt.subplots()
x_index = 3
y_index = 0
colors = ['blue', 'red', 'green']
for label, color in zip(range(len(iris.target_names)), colors):
ax.scatter(iris.data[iris.target==label, x_index],
iris.data[iris.target==label, y_index],
label=iris.target_names[label],
c=color)
ax.set_xlabel(iris.feature_names[x_index])
ax.set_ylabel(iris.feature_names[y_index])
ax.legend(loc='upper left')
plt.show()
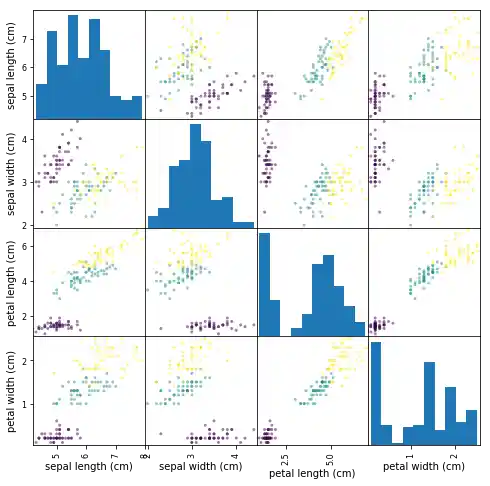
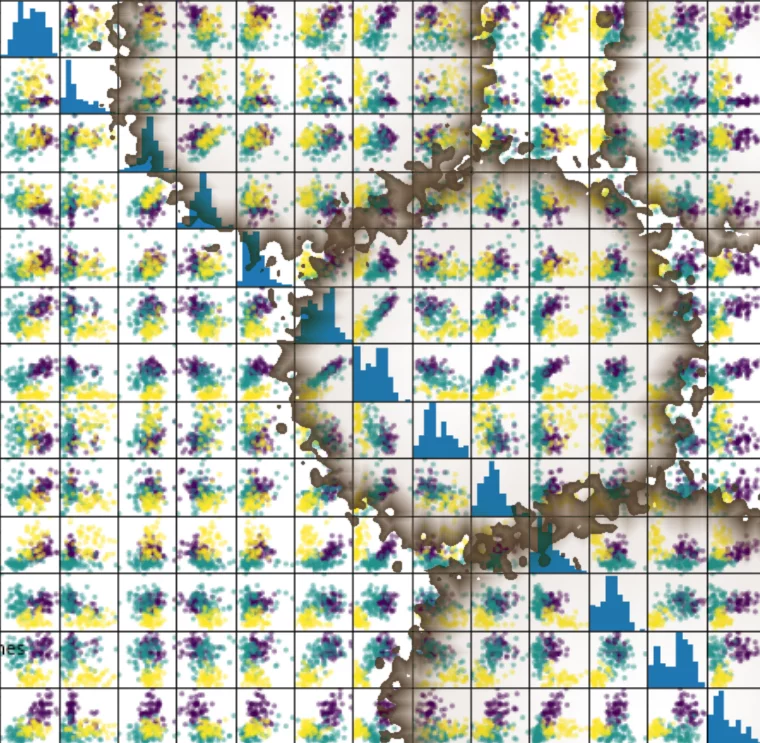
Streudiagramm Matrix¶
Anstatt die Daten jeweils als einzelne Plots zu betrachten, können wir ein gängiges von Analysten verwendetes Tool vewenden. Es erzeugt eine Streudiagramm-Matrix (Scatterplot-Matrix).
Streudiagramm-Matrizen zeigen Streudiagramme zwischen allen Features im Datensatz sowie Histogramme, um die Verteilung der einzelnen Features anzuzeigen.
import pandas as pd
iris_df = pd.DataFrame(iris.data, columns=iris.feature_names)
pd.plotting.scatter_matrix(iris_df,
c=iris.target,
figsize=(8, 8)
);
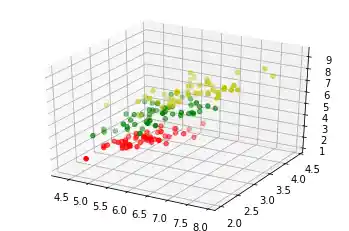
3-Dimensionale Visualisierung¶
import matplotlib.pyplot as plt
from sklearn.datasets import load_iris
from mpl_toolkits.mplot3d import Axes3D
iris = load_iris()
X = []
for iclass in range(3):
X.append([[], [], []])
for i in range(len(iris.data)):
if iris.target[i] == iclass:
X[iclass][0].append(iris.data[i][0])
X[iclass][1].append(iris.data[i][1])
X[iclass][2].append(sum(iris.data[i][2:]))
colours = ("r", "g", "y")
fig = plt.figure()
ax = fig.add_subplot(111, projection='3d')
for iclass in range(3):
ax.scatter(X[iclass][0], X[iclass][1], X[iclass][2], c=colours[iclass])
plt.show()


 Buch kaufen
Buch kaufen
 Buch kaufen
Buch kaufen
 Buch kaufen
Buch kaufen